單細胞轉錄組長讀長測序揭示癌癥研究中難以獲得的重要見解
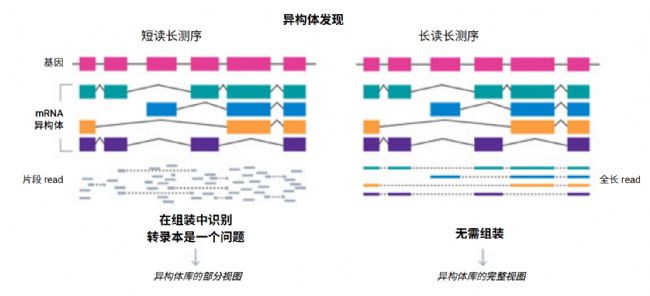
癌癥研究中,在異構體水平上對單細胞轉錄組進行特征分析的能力至關重要。常規單細胞轉錄組學中,短讀長序列僅能捕獲基因水平的信息。
Iso-Seq® 技術(Isoform Sequencing,PacBio® HiFi 測序的全長 RNA 測序技術)已被證實可推動癌癥研究,該技術能在單細胞水平上對異構體進行明確的特征分析,而其他長讀長技術則缺乏識別獨特分子標識符(UMI)和細胞條形碼(CBC)所需的準確性。PacBio Kinnex™ RNA 試劑盒以 MAS-Seq 技術為基礎(該技術可將 cDNA 等擴增子連接成更長的片段),進一步提升了 HiFi 長讀長測序儀的測序通量。對連接分子進行測序所產生的 HiFi 讀長序列,可通過生物信息學方法拆分,從而獲取原始的 cDNA 序列。
本文重點介紹幾篇將 Iso-Seq 技術應用于癌癥研究的文獻,闡釋單細胞 Iso-Seq 數據如何用于檢測融合基因、識別可能作為疫苗候選新表位的新型異構體,以及追溯克隆進化過程。
檢測融合基因
短讀長測序可輕松識別融合連接點,但由于讀長不足,無法完整解析單個融合轉錄本的結構。Dondi et al.(2023)將 Kinnex 連接法的早期版本應用于卵巢癌樣本的單細胞全長 RNA 測序中。在他們的 Kinnex 測序數據中,鑒定出了一種患者特異性的 IGFBP2::TESPA1 融合基因;而在匹配的短讀長測序數據中,該融合基因此前被誤判為 TESPA1 基因高表達,因為短讀長測序僅捕獲到了該融合基因的 3' 端融合部分,即 TESPA1 基因部分。
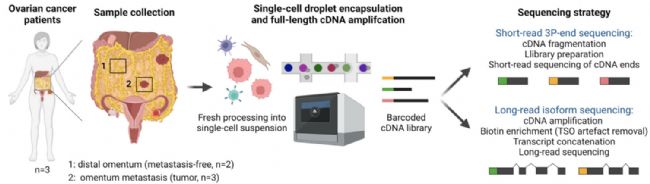
圖1. 對新鮮處理的高級別漿液性卵巢癌(HGSOC)大網膜轉移灶,以及與患者匹配的無腫瘤遠端大網膜組織活檢樣本進行的單細胞 RNA 短讀長及長讀長測序。
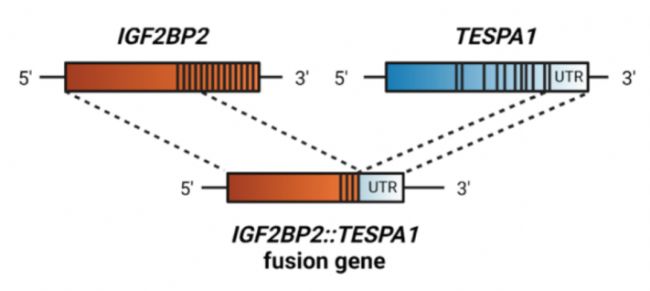
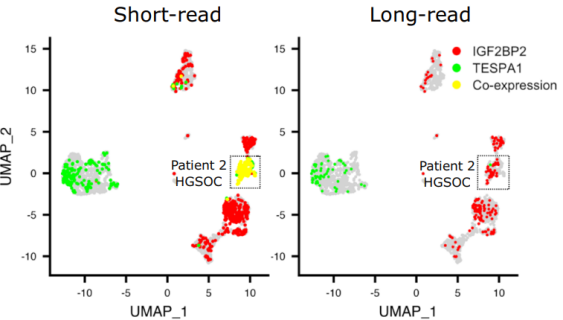
圖2. 長讀長單細胞 RNA 測序鑒定出腫瘤特異性融合基因 IGF2BP2::TESPA1,而短讀長數據將該基因誤判為 TESPA1 過表達。
腫瘤新抗原發現 - 新型異構體發現在轉化研究中的應用
剪接失調(splicing dysregulation)是大多數癌癥中普遍存在的特征,但人們對異常剪接的整體情況仍知之甚少。Li et al.(2024)的研究中,研究人員通過單細胞 RNA 測序技術研究結直腸癌(colorectal cancer, CRC),在腫瘤上皮細胞中鑒定出 394 種失調的轉錄本結構。該研究同時發現了腫瘤特異性及異構體特異性的 RNA 編輯事件,通過質譜驗證篩選出具有腫瘤特異性的新型異構體。研究人員開發了一種算法,依據人類白細胞抗原(HLA)結合親和力對這些潛在的新抗原進行排序。借助這一方法,他們發現了 4 種新型腫瘤特異性異構體,這些異構體對廣大結直腸癌患者群體均具有較強的 HLA 結合親和力,有望成為癌癥疫苗的候選分子。
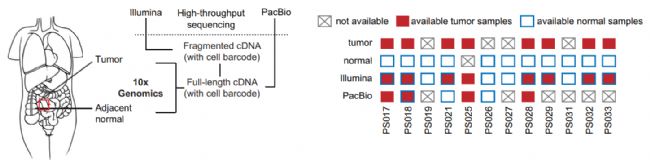
圖3. 工作流程示意圖:12 例結直腸癌(CRC)患者腫瘤樣本與正常樣本的長讀長(PacBio)及短讀長(Illumina)單細胞 RNA 測序(scRNA-seq)。
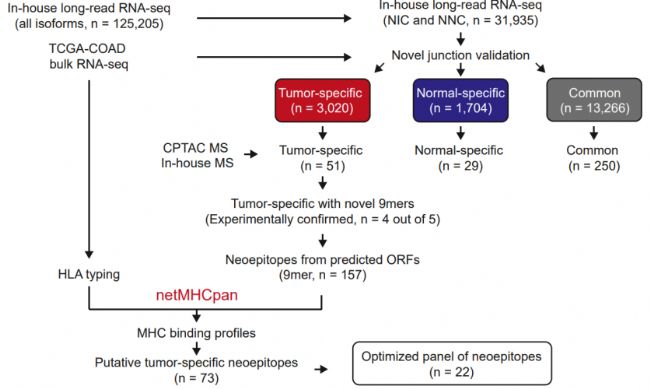
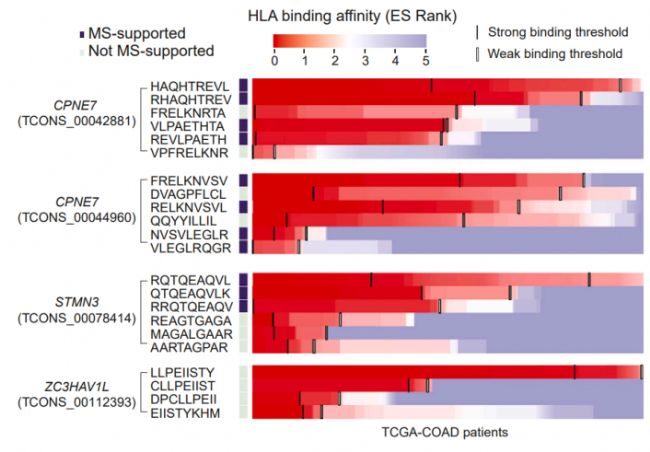
圖4. 上圖為用于癌癥疫苗研發的、基于新型腫瘤特異性反復出現的轉錄本異構體的新表位鑒定流程;下圖展示22 個新表位針對 TCGA-COAD 患者 HLA 等位基因的親和力及排序。
追蹤克隆進化
常規短讀長單細胞 RNA 測序(short-read single-cell RNA-Seq)僅能覆蓋轉錄本的 5' 端或 3' 端,因此在基于腫瘤體細胞突變確定細胞基因型的能力上存在局限。相比之下,單細胞 Iso-Seq 技術能夠對來自多個單細胞平臺的全長 cDNA 進行測序。
Black et al.(2024)將PacBio Kinnex單細胞RNA試劑盒應用于由 10x Genomics Chromium平臺生成的單細胞 cDNA 中,該cDNA來源于攜帶BTK耐藥突變的慢性淋巴細胞白血病(CLL)樣本,研究表明應用長讀長測序結果在基因分型能力方面有顯著提升。對于在≥1% 的細胞中存在的變異位點,Illumina短讀長測序僅覆蓋了其中的7.61%,而Kinnex數據則覆蓋了18.59%的此類變異位點。這一結果進而幫助優化了其中一個樣本的亞克隆結構,清晰呈現出兩個不同的突變亞克隆 —— 這兩個亞克隆此前被錯誤地識別為單一亞克隆。
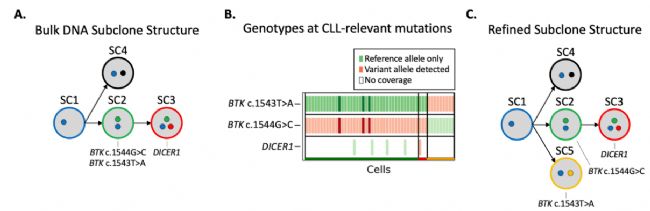
圖5. A) 從批量 DNA 測序數據中識別出的亞克隆結構。慢性淋巴細胞白血病(CLL)相關基因突變會標注在其所在的亞克隆下方。B) 患復發樣本的基因型矩陣圖有助于完善原始亞克隆結構。綠色表示在該細胞內特定變異位點的scRNA-seq中僅存在參考等位基因;紅色表示該細胞內至少有一個scRNA-seq測序讀數包含體細胞變異等位基因。顏色越深,表明支持該基因型的reads數量越多。為提高區分亞克隆的分辨率,僅納入了CLL相關基因突變。C) 經完善的亞克隆結構顯示,包含BTK基因 c.1543T>A突變的亞克隆,與包含BTK基因c.1544G>C 突變和DICER1基因突變的亞克隆相互獨立。
Wedemeyer et al.(2024)研究發現,當使用Kinnex單細胞RNA試劑盒對一株攜帶已知單核苷酸變異(PIK3CA:c.3139C>T)的患者來源成纖維細胞進行測序時,長讀長測序在15.3%的細胞中覆蓋了該變異位點;相比之下,使用Illumina短讀長測序時,僅在1.2%的細胞中覆蓋了該變異位點。此外,通過將突變型細胞與野生型細胞進行對比,研究人員發現了通常與上皮-間質轉化(epithelial-mesenchymal transition,EMT)相關的獨特基因表達模式及信號通路,這一發現為理解為何僅存在于少量細胞群體中的變異,能導致巨腦-毛細血管畸形綜合征(MCAP)患者出現顯著臨床表型提供了思路。
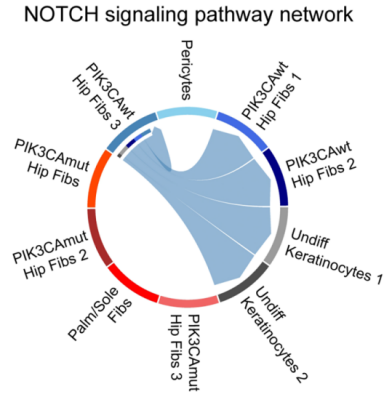

圖6. 上圖Chord diagram顯示,在野生型 PIK3CA(PIK3CAwt)成纖維細胞與未分化角質形成細胞之間存在Notch介導的信號傳導,而在突變型PIK3CA(PIK3CAmut)成纖維細胞與未分化角質形成細胞之間則無此信號傳導。下圖展示野生和突變型細胞富集前10的Gene Ontology BP點圖。
小結
單細胞轉錄組長讀長測序(Iso-Seq 技術),能夠揭示癌癥研究中難以通過短讀長測序獲得的重要見解。單細胞 Iso-Seq技術還可結合 Kinnex 試劑盒應用,提高測序通量,用于檢測新型異構體、融合轉錄本和新抗原,同時對單細胞進行基因分型并追蹤克隆群體,最終獲得對癌癥轉錄組更全面的認識。
北京怡美通德科技發展有限公司是10x Genomics和PacBio的官方授權代理商,代理美國10x Genomics公司生產的Chromium單細胞測序建庫系列產品、Visium空間轉錄組系列產品以及Xenium組織空間原位分析系統;美國PacBio公司生產的Vega桌面式三代測序系統,提供儀器配套的試劑耗材供貨、產品演示實驗、產品安裝、使用培訓以及售前售后技術咨詢、生物信息學分析等相關技術服務。
如果您想了解更多關于10x Genomics和PacBio的信息,歡迎掃描下方二維碼或點擊文末左下角的“閱讀原文”填寫信息,我們會在第一時間與您聯系!

參考文章:
1.Black et al. (2024). Long-read single-cell RNA sequencing enables the study of cancer subclone specific genotype and phenotype in chronic lymphocytic leukemia. BioRxiv. https://doi.org/10.1101/2024.03.15.585298
2.Dondi, A., Lischetti, U., et al. (2023). Detection of isoforms and genomic alterations by high-throughput full-length single-cell RNA sequencing in ovarian cancer. Nature Communications, 14(1), 7780. https://doi.org/10.1038/s41467-023-43387-9
3.Li, Z., Zhang, B., Chan, J. J., et al. (2024). An isoform resolution transcriptomic atlas of colorectal cancer from long-read single-cell sequencing. Cell Genomics, 4(9). https://doi.org/10.1016/j.xgen.2024.100641
4.Wedemeyer, M. A., et al. (2024). Defining the transcriptome of PIK3CA-altered cells in a human capillary malformation using single cell long-read sequencing. Scientific Reports, 14(1), 25440. https://doi.org/10.1038/s41598-024-72167-8