10x GEM-X試劑單細胞CRISPR篩選實驗方案流程及應用
CRISPR(Clustered regularly interspaced short palindromic repeats)是細菌和古菌所使用的一種適應性免疫系統,其通過記錄和靶向病毒的 DNA 序列來抵御入侵的病毒。這一機制已被重新改造為一種簡單、可靠且用途廣泛的技術,用于哺乳動物及其他生物的基因組編輯,使研究人員能夠對各類生物學過程和疾病狀態進行研究。
CRISPR篩選是功能基因組學研究人員不可或缺的工具,但傳統篩選方法既復雜又耗時,而且數據輸出的分辨率低。傳統篩選實驗使用Bulk RNA 輸入來評估所有細胞的平均基因表達水平,從而掩蓋了細胞異質性;與之不同的是,單細胞 CRISPR 篩選技術能在單細胞水平上,評估整個轉錄組中多個基因以及每個單獨 sgRNA 的擾動效應。(圖1)
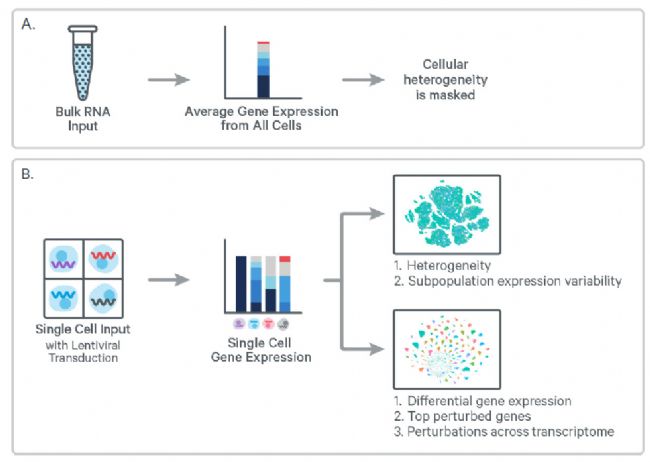
圖1
如果說傳統CRISPR是一把分子剪刀,那么單細胞CRISPR可謂是一把多功能的瑞士軍刀,可快準狠的破解生物的多重奧秘。
來自單細胞分析引領品牌10x Genomics的Chromium單細胞CRISPR篩選技術帶來了一個更簡單、更快速的工作流程,采用Feature Barcode技術和新一代測序技術來檢測單鏈向導RNA(sgRNA),該技術可評估慢病毒向導 RNA(sgRNA)轉導的單細胞輸入,提供了一種高通量、可擴展的方法,能在同一個單細胞中同時獲取基因表達譜和 CRISPR 介導的擾動表型。(圖2)
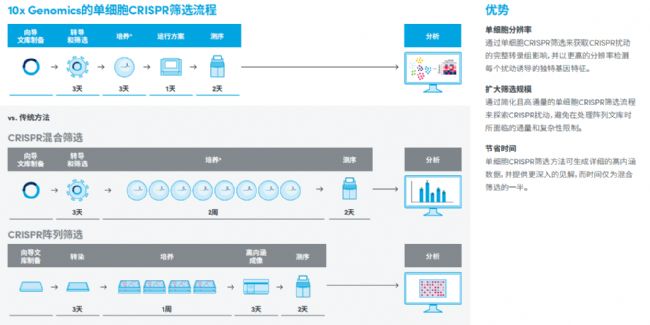
圖2 * 包括擾動或施加刺激后的培養時間
10x Genomics最新版GEM-X試劑提供了兩種單細胞CRISPR篩選實驗方案:應用于新鮮樣本的GEM-X 5’ CRISPR Screening,以及應用于固定后樣本的GEM-X Flex CRISPR Screening。這兩款產品均能將單細胞基因表達分析與CRISPR篩選相結合,以單細胞分辨率將CRISPR擾動與完整的基因表達表型直接關聯。
GEM-X 5’ CRISPR Screening
采用 Feature Barcode 技術進行 CRISPR 篩選的 Chromium 單細胞免疫組庫分析(也稱為單細胞 5' 端 CRISPR 篩選)提供了一種多組學研究方法,可從同一個單細胞中同時檢測基因表達、CRISPR 向導分子、細胞表面蛋白和 / 或免疫細胞克隆型頻率。這種單細胞 5' 端 CRISPR 篩選方法與大多數 Cas9 CRISPR 載體兼容,且無需在向導 RNA(sgRNA)中整合特定捕獲序列。
10x Genomics建議每個靶向向導 RNA(sgRNA)使用100-200個細胞,這能確保有足夠的統計檢驗力來判斷擾動的顯著性。對于非靶向向導 RNA(sgRNA),其對于提供計算擾動所需的基線至關重要,建議使用 5-10% 的非靶向sgRNA。Chromium 單細胞 CRISPR 篩選檢測試劑盒Chromium GEM-X 5’ 芯片的每個通道上可回收多至 20,000個細胞,其中1,000-2,000個(5%-10%)細胞為含非靶向向導 RNA(sgRNA)的細胞。每個靶向向導 RNA(sgRNA)應對應 100-200 個細胞。采用這種設置,使用GEM-X 5’ 芯片的單個通道即可檢測約 90-180 個向導 RNA(sgRNA)。如果您希望檢測更多種sgRNA,可加大細胞量,上滿芯片所有8個通道且回收160,000 個細胞,便可進行Pooled CRISPR篩選,檢測約1000個sgRNA。
【實驗流程】
1. 確認sgRNA兼容性
為兼容 Chromium 單細胞 5' 端 CRISPR 篩選檢測試劑盒,向導 RNA(sgRNA)需經工程改造以適配標準 Cas9 系統,圖3 顯示了5’ CRISPR 反轉錄引物的結合區域。
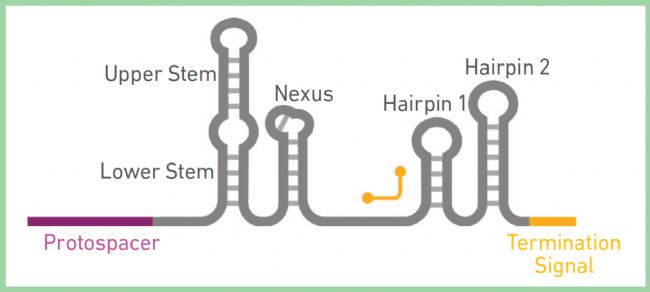
圖3
CRISPR 逆轉錄(RT)引物設計于向導 RNA(sgRNA)骨架的高度保守區域,以實現對各類Cas9系統的高兼容性。該序列(5’-CAAGTTGATAACGGACTAGCC-3’)與向導 RNA 骨架序列互補。在進行單細胞CRISPR實驗前,建議將CRISPR 引物序列的反向互補序列,與目標 sgRNA 向導文庫進行 BLAST 比對,以確認兼容性。上述引物序列的反向互補序列為:5'-GGCTAGTCCGTTATCAACTTG-3'。若向導 RNA 骨架不含上述序列,可設計定制化富集引物來擴增特殊向導 RNA。
2.sgRNA捕獲及文庫構建
將帶有條形碼的單細胞 5'凝膠珠、含有轉導細胞的 Master Mix(混合反應液)以油(Partitioning Oil)GEM-X 5’ 芯片上混合,即可生成 GEMs(油包水微滴)。GEMs 生成后,凝膠珠立即溶解,油包水內細胞隨之裂解。凝膠珠釋放出寡核苷酸,與細胞裂解物以及含有逆轉錄(RT)試劑和引物混合物的 Master Mix 混合。通過孵育 GEMs,可同時從細胞mRNA 中生成帶有 10x 條形碼的全長 cDNA,并從 sgRNA中生成帶有條形碼的 DNA,進而可從這些帶有 10x 條形碼的 cDNA 中構建出可用于測序的基因表達文庫和 CRISPR 篩選文庫(圖4)。
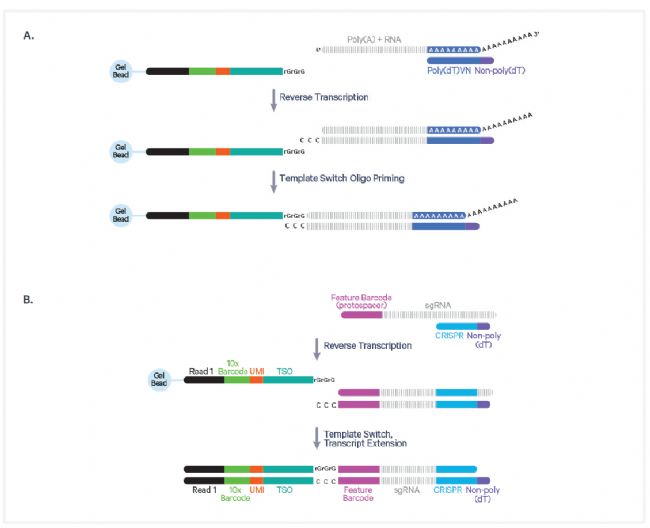
圖4
3.文庫測序及數據分析
在確定特定擾動的效應時,目標基因表達的動態測序深度范圍是關鍵因素。由于特定基因的 UM數量取決于基因表達文庫的整體測序深度,文庫的測序深度越深,可檢測到的 UMI 就越多(直至基因表達文庫達到飽和狀態,此時每增加一次新的測序讀數,檢測到新 UMI 的可能性極低)。特定基因檢測到的 UMI 數量越多,其表達變化被檢測到且具有顯著性的可能性就越大。下圖(圖5)建議了5’表達譜、5’ CRISPR等文庫的最低測序量。

圖5
使用10x Genomics數據分析軟件Cell Ranger 9.0.0版本分析每個反應通道的數據,如實驗中大量細胞上樣到多個反應通道,可使用cellranger aggr流程將生成的多個輸出結果匯總在一起。
GEM-X Flex CRISPR Screening
10x Genomics Chromium GEM-X Flex基因表達分析技術為固定樣本中的基因表達檢測提供了全面且可擴展的解決方案。在該檢測中,10x Genomics公司提供了預先設計的人和小鼠的全轉錄組探針panel,用于目標基因雜交。您可根據本文件中的指導設計定制化探針,添加到商品化的探針試劑盒中,用于在檢測基因表達的同時檢測CRISPR 篩選。
1. CRISPR Guide Probe設計合成及使用說明
用于 GEM-X Flex 基因表達分析的 CRISPR 向導探針設計請見圖6概述,探針設計的序列詳見表1,所使用的 CRISPR LHS 探針 barcode 序列(CR001-CR016)列于表 2 中。
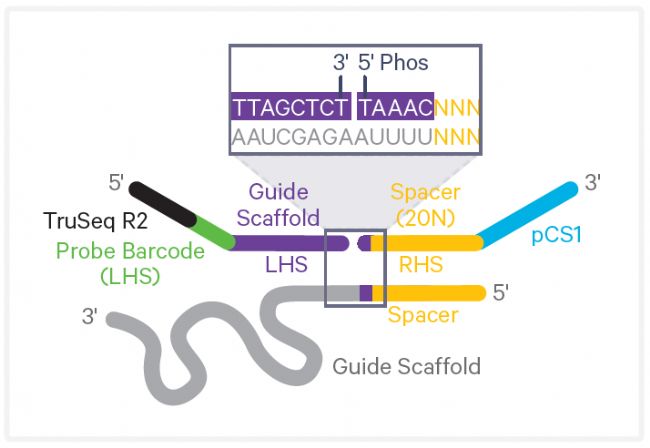
圖6
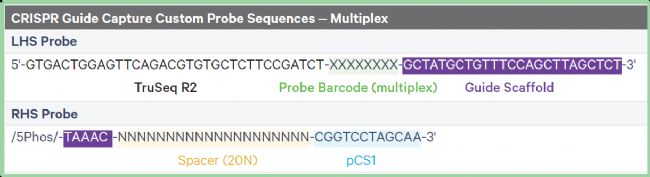
表1
要設計用于GEM-X Flex檢測的CRISPR向導捕獲定制探針,必須預先知曉向導骨架(guide scaffold)和間隔序列(spacer sequences),探針設計參考以下原則:
· 左側探針(LHS Probe)包含TruSeq R2、探針條形碼(Probe Barcode)以及一段25 bp的向導骨架互補序列。TruSeq 接頭可添加CRISPR文庫的Index。
· CRISPR左側探針條形碼(LHS Probe Barcode)序列與全轉錄組分析(WTA)探針中使用的序列不同,詳見表 2。
· 右側探針(RHS Probe)包含 5’磷酸基團(/5Phos/)、一段 5 bp 的向導骨架互補序列、一段20 bp的靶向間隔序列互補序列,以及部分Capture Sequence 1(pCS1)。
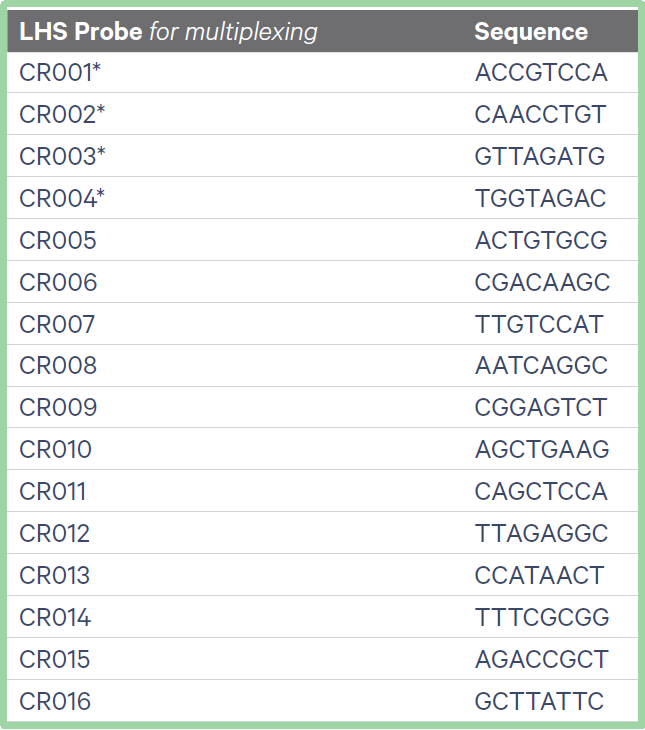
表2
在多樣本混樣實驗中,每個左側探針(LHS Probe)必須帶有唯一的 8 堿基探針條形碼(CR001-CR016;序列詳見表 2),且每個雜交反應的 CRISPR 探針和全轉錄組擴增(WTA)探針均需使用唯一的探針條形碼對,即同一 CRISPR 探針條形碼不得與多個 WTA 探針條形碼搭配使用。在單重實驗中,可省略該探針條形碼。
定制探針可從任何寡核苷酸合成供應商處訂購。10x Genomics已在IDT提供的多種形式定制探針中進行了測試,包括DNA寡核苷酸(標準脫鹽)、Ultramer DNA 寡核苷酸和 oPool 寡核苷酸庫。在有限的測試中,所有形式均觀察到了可比較的結果。
合成探針關鍵指導原則如下:
· 探針應經過標準脫鹽處理。
· 無需進行 HPLC 純化。
· 探針應重懸于 IDTE(或低 EDTA TE 緩沖液)中。
· 右側(RHS)探針必須進行 5' 磷酸化。
· 帶條形碼的左側(LHS)探針應單獨訂購,采用標準脫鹽形式。
· 右側(RHS)探針可單獨以標準脫鹽形式訂購,也可作為 oPool 寡核苷酸庫訂購。
使用CRISPR Guide Probe,需首先制備一個混合的 spike-in 庫,其中包含終濃度為 40 nM 的各右側(RHS)探針,以及終濃度為400 nM的帶條形碼的CRISPR 左側(LHS)探針。在向重懸的細胞沉淀中加入10x Genomics的人/小鼠全轉錄組擴增(WTA)探針后,再向樣本中加入2.5 μl該spike-in庫。
每個靶向sgRNA建議上樣100–200個細胞,以確保有足夠的統計效力來確定擾動的顯著性。對于非靶向 sgRNA(其對提供計算擾動的基線至關重要),建議上樣500–1000個細胞。當使用全部16種探針進行多重檢測時,使用GEM-X Flex檢測可從每個反應可回收多達320,000個細胞。當使用芯片的全部8個孔(目標回收率約為2.5×10⁶個細胞)時,可檢測的sgRNA總數(包括非靶向 sgRNA)為12,000–25,000個。
2.實驗流程及需要準備的額外試劑
GEM-X Flex基因表達和CRIPSR檢測的實驗流程如下圖(圖7)
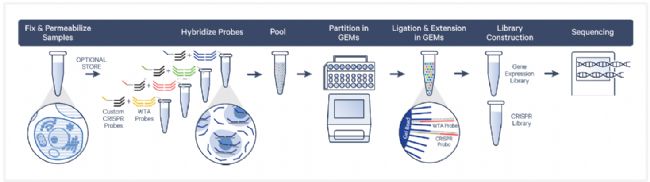
圖7
使用GEM-X Flex檢測試劑盒進行CRISPR篩選時,除GEM-X Flex試劑盒外,還需要以下額外的10x Genomics試劑:
・Dual Index Kit TT Set A 96 rxns, PN-1000215
・Amp Mix,可從GEM-X Single Cell 5' Feature Barcode Kit (PN-1000703)中獲取。
・用戶自備的 TruSeq 引物,引物應訂購標準脫鹽級別的產品。
- 正向引物(TruSeq Read 1):CTACACGACGCTCTTCCGATCT
- 反向引物(TruSeq Read 2):GTGACTGGAGTTCAGACGTGTG
為進行 CRISPR 篩選,需要在原User Guide(CG000789)基礎上對以下步驟進行修改或新增,具體操作請參考10x Technical Note CG000814:
Step 1: Probe Hybridization (Modified)
Step 4: Pre-Amplification PCR (Modified)
New Step: Feature PCR
New Step: CRISPR Screening Library Construction
3.文庫測序及數據分析
建議將GEM-X Flex CRISPR 篩選文庫與基因表達文庫混合二者混合,以在測序過程中維持核苷酸多樣性。GEM-X Flex 基因表達文庫的建議最低測序深度為 10,000 read Pairs / 細胞,GEM-X Flex CRISPR 篩選文庫的建議最低測序深度為 5,000 read pairs / 細胞。圖8和圖9 分別展示了兩文庫結構及測序讀長建議。
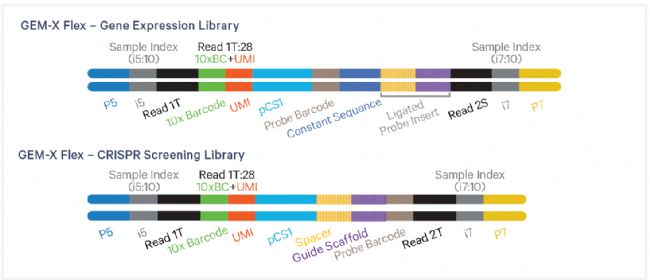
圖8

圖9
10x Genomics數據分析軟件Cell Ranger支持對單重和多重 Flex+CRISPR 數據的分析。在分析多重實驗時,Multi Config CSV 文件需要同時指定 WTA(全轉錄組擴增)和 CRISPR 的探針條形碼,詳情請參見 10x Genomics 支持網站(Flex Gene Expression & Feature Barcode Analysis with Cell Ranger multi (Singleplex & Multiplex) | 10x Genomics)。與目前市場上的 CRISPR 工作流程類似,需要使用特征參考文件(feature reference file)來識別不同的前間區序列鄰近基序(protospacer)。請參照 “5' CRISPR 向導捕獲” 的特征參考示例進行操作(Cell Ranger Feature Reference CSV | 10x Genomics)。
北京怡美通德科技發展有限公司是10x Genomics的官方授權代理商,代理美國10x Genomics公司生產的Chromium單細胞測序建庫系列產品、Visium空間轉錄組系列產品以及Xenium組織空間原位分析系統,提供儀器配套的試劑耗材供貨、產品演示實驗、產品安裝、使用培訓以及售前售后技術咨詢、生物信息學分析等相關技術服務。
如果您想了解更多關于10x Genomics的信息,歡迎掃描下方二維碼或點擊文末左下角的“閱讀原文”填寫信息,我們會在第一時間與您聯系!