文獻解讀:評估代謝RNA標記技術在高通量單細胞RNA測序中的應用

期刊:Nature communications
影響因子:15.7
主要技術:華大C4 scRNA-seq,10x scRNA-seq
導語
代謝RNA標記結合高通量單細胞RNA測序(scRNA-seq)能夠精確測量復雜生物過程中基因表達動態,如細胞狀態轉換和胚胎發育。該技術通過誘導堿基轉換標記新合成的RNA以進行檢測,依賴于轉換效率、RNA完整性和轉錄本回收率。這些因素受所選化學轉換方法和平臺兼容性的影響。本研究使用Drop-seq平臺對十種化學轉換方法進行測試,分析了52,529個細胞。分析發現,基于beads的方法,特別是mCPBA/TFEA,優于原位(In-suit)方法。為評估體內應用,作者將這些優化方法應用于9,883個斑馬魚胚胎細胞在母體到合子轉換期間,識別并驗證了合子激活轉錄本,增強了合子基因檢測能力。此外,作者還評估了兩種捕獲效率更高的商業化平臺,發現固相上的碘乙酰胺(IAA)化學方法最為有效。本研究結果為選擇最佳化學方法和scRNA-seq平臺提供了關鍵指導,推動了復雜生物系統中RNA動態研究。
主要技術
華大C4 scRNA-seq,10x scRNA-seq
研究結果
1. 實驗設計和計算質量控制評估
為全面測試代謝標記單細胞RNA測序(scRNA-seq)中不同化學轉化方法的性能,作者以Drop-seq平臺為基礎,在源自斑馬魚胚胎的ZF4成纖維細胞系上測試了10種化學轉化方法,通過dynast流程及專用質控流程,從RNA完整性、轉化效率、RNA回收率三個維度評估方法優劣。結果顯示,mCPBA/TFEA(pH5.2)等磁珠轉化方法表現最優,平均T-to-C替換率超8%,且對cDNA完整性影響較小,每個細胞在10,000條測序reads下可檢測約2,044個基因和5,468個UMI,與未處理樣本水平相當,且磁珠方法中T-to-C替換率是原位轉化方法的 2.32倍。這一結果為代謝標記scRNA-seq中化學轉化方法的選擇提供了直接基準,明確了磁珠上轉化方法在效率和完整性上的優勢,為后續研究奠定方法學基礎。
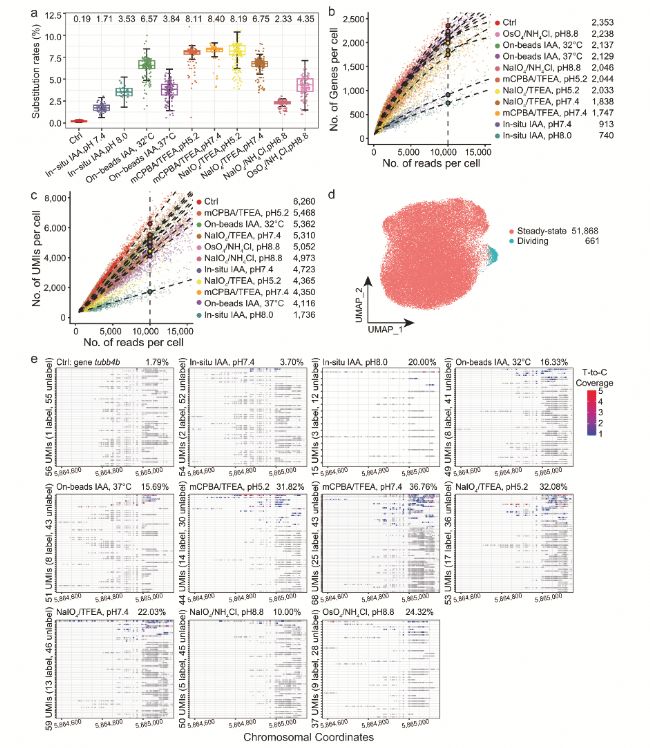
圖1 使用ZF4細胞對十種化學轉化方法進行比較和評估。
2. 細胞周期中的 mRNA 控制策略
為探究代謝RNA標記在細胞周期mRNA調控研究中的應用價值,作者將52,529個經不同化學轉化處理的細胞分為穩態和分裂細胞集群,結合流式細胞術、相關性分析及基因表達分析評估方法效果。結果表明,4sU標記本身不影響細胞周期分布,但OsO₄和IAA(37°C)處理會導致分裂細胞減少;磁珠上轉化方法在標記與未標記RNA檢測上一致性更高(Pearson’sr>0.9),且mCPBA類方法能高效識別細胞周期相關基因(如tubb4b),其標記轉錄本比例與T-to-C替換率正相關,同時細胞周期基因的RNA半衰期顯著短于管家基因。這一發現證實了優化后的代謝標記方法可準確捕捉細胞周期中的RNA動態,為解析細胞周期相關的基因表達調控機制提供了可靠技術手段,也為后續研究RNA穩定性與細胞功能的關聯提供了數據支持。
3. 鑒定斑馬魚胚胎發育中的合子激活轉錄本
為驗證優化化學轉化方法在體內的適用性,研究以斑馬魚母源-合子轉換為模型,在5.5hpf胚胎細胞(合子轉錄本占比9.33%)中應用不同方法,通過新RNA與總RNA比例(NTR)劃分母源、合子及母源-合子基因,并與公開數據集對比。結果顯示,基于mCPBA的方法能識別更多合子基因,約78%新轉錄RNA基因為母源-合子基因(MZ),與斑馬魚bulkRNA-seq及爪蟾新生RNA-seq結果高度一致,且T-to-C替換率越高,高NTR閾值下合子基因識別差異越顯著,全胚胎原位雜交及內含子探針驗證進一步確認了合子轉錄本的準確性。這一成果證明優化方法可高效解析體內胚胎發育中的RNA動態,為深入研究母源-合子轉換機制及合子基因組激活(ZGA)提供了關鍵技術支撐,也為其他物種早期胚胎發育的轉錄組研究提供了參考。
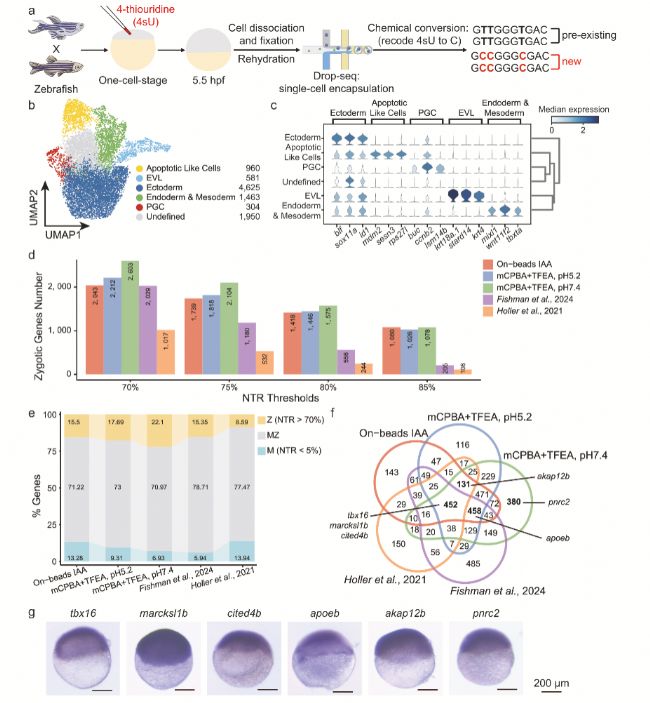
圖2 利用改進的化學轉化方法在斑馬魚胚胎發育中鑒定合子激活轉錄本
4.鑒定斑馬魚胚胎發育中的合子激活轉錄本
為解決Drop-seq平臺(細胞捕獲效率約5%)在稀有細胞樣本研究中的局限性,研究對比了10×Genomics和華大C4兩種高捕獲效率(約50%)商業化平臺,測試不同化學轉化方法與平臺的兼容性。結果顯示,華大C4支持磁珠上轉化,其結合IAA(32°C)方法的T-to-C替換率最高(8.44%),文庫復雜度也更優;10×Genomics僅限原位轉化,轉化效率較低但文庫復雜度較高;相同原位化學條件下,C4平臺的T-to-C替換率(5.74%)高于其他平臺,且商業化平臺在基因和UMI檢測數上均優于定制化Drop-seq。這一對比明確了不同平臺的優勢與適用場景,為稀有細胞(如早期胚胎細胞)研究提供了平臺與方法的最優組合方案,有助于推動低細胞量樣本的轉錄組動態研究。
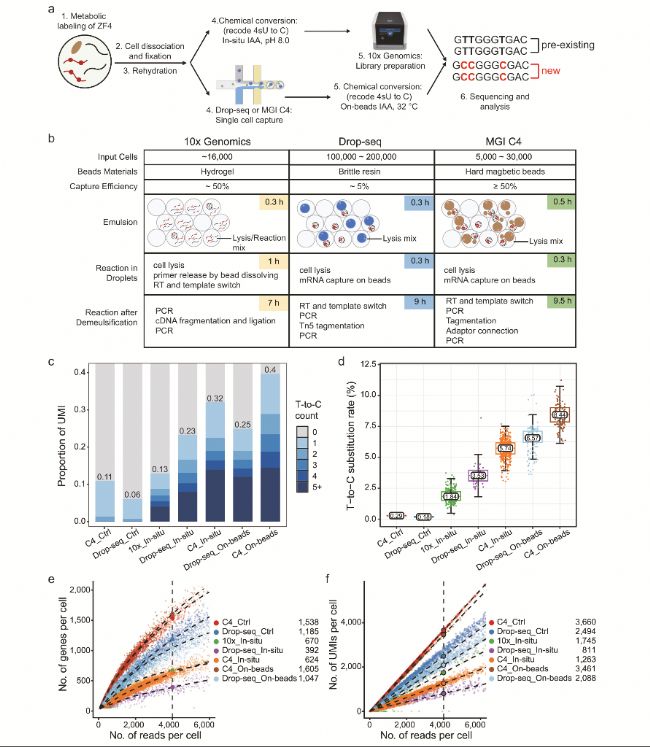
圖3 10×Genomics、Drop-seq和華大C4高通量單細胞平臺的比較
參考文獻:
[1]ZhangX,PengM,ZhuJ,ZhaiX,WeiC,JiaoH,WuZ,HuangS,LiuM,LiW,YangW,MiaoK,XuQ,ChenLandHuP2025BenchmarkingmetabolicRNAlabelingtechniquesforhigh-throughputsingle-cellRNAsequencingNatCommun165952.