利用人工智能驅動的空間蛋白質組學繪制腫瘤免疫圖譜
采用人工智能驅動的空間蛋白質組學分析方法研究小鼠癌癥模型中的腫瘤-免疫相互作用
 未經治療腫瘤的空間圖譜分析可呈現腫瘤免疫結構的整體特征,有助于理解治療反應。具有免疫活性的小鼠模型對于識別腫瘤發生發展過程中免疫依賴性事件至關重要。要表征這些具有完整免疫系統及相互作用細胞組分的模型,需要采用多重標記分析技術。我們展示了一種基于人工智能的空間蛋白質組學方法,用于研究小鼠癌組織中的腫瘤-免疫互作機制。
未經治療腫瘤的空間圖譜分析可呈現腫瘤免疫結構的整體特征,有助于理解治療反應。具有免疫活性的小鼠模型對于識別腫瘤發生發展過程中免疫依賴性事件至關重要。要表征這些具有完整免疫系統及相互作用細胞組分的模型,需要采用多重標記分析技術。我們展示了一種基于人工智能的空間蛋白質組學方法,用于研究小鼠癌組織中的腫瘤-免疫互作機制。
01 多重成像與人工智能分析的整合應用
本研究探討了癌癥進展與治療過程中免疫系統與腫瘤細胞之間復雜的相互作用關系。Cell DIVE 多重成像技術能在單張組織切片上檢測數十種生物標志物,提供展示各類細胞類型的全組織分子圖譜。Cell Signaling Technology 提供的 IHC 驗證抗體與 Cell DIVE 完美兼容,可增強組織樣本中各類細胞的檢測與表型分析。Aivia 軟件的整合通過 AI 引導的分割、表型分析和聚類分析簡化了空間生物學工作流程。以 4T1 小鼠腫瘤模型為例的研究證實了該空間蛋白質組學工作流程的實用性,揭示了腫瘤內部異質性免疫景觀及復雜的空間關系。
通過 Aivia 平臺進行 AI 輔助分割與表型分析,我們對 Cell DIVE 多重全組織圖像中所有標志物的表達特征進行了系統表征(圖 1)。
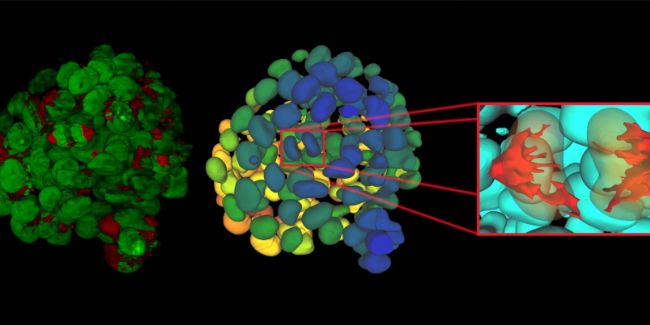
 圖 1:使用 Aivia 進行多重圖像分析。A. Cell DIVE 全組織多重圖像及局部放大面板。B. 通過 Aivia 的多重細胞檢測方案實現細胞膜與核膜分割,將各細胞識別為獨立對象。C. AI 輔助表型分析鑒定各類細胞,包括免疫細胞、腫瘤細胞、成纖維細胞、內皮細胞及 Glut1 陽性細胞(可能屬于免疫或腫瘤細胞等不同類型)。
圖 1:使用 Aivia 進行多重圖像分析。A. Cell DIVE 全組織多重圖像及局部放大面板。B. 通過 Aivia 的多重細胞檢測方案實現細胞膜與核膜分割,將各細胞識別為獨立對象。C. AI 輔助表型分析鑒定各類細胞,包括免疫細胞、腫瘤細胞、成纖維細胞、內皮細胞及 Glut1 陽性細胞(可能屬于免疫或腫瘤細胞等不同類型)。
02 腫瘤微環境對免疫細胞分布的影響
研究揭示了特定免疫細胞類型(如 T 細胞、巨噬細胞和樹突狀細胞)相對于腫瘤內缺氧與壞死區域的空間分布特征,表明腫瘤微環境對免疫細胞行為的影響及其對治療結局的潛在意義。
在同基因 4T1 小鼠腫瘤模型中,利用 Cell DIVE 和 Aivia 進行 AI 驅動的空間蛋白質組學分析,揭示了一個復雜多樣的免疫圖譜。總體而言,這項研究強調了多重成像技術與 AI 驅動分析相結合的重要性,以理解腫瘤微環境內的空間關系及相互作用,為開發更有效的癌癥療法提供了見解。
相關產品

 AI圖像分析軟件 Aivia
AI圖像分析軟件 Aivia
 未經治療腫瘤的空間圖譜分析可呈現腫瘤免疫結構的整體特征,有助于理解治療反應。具有免疫活性的小鼠模型對于識別腫瘤發生發展過程中免疫依賴性事件至關重要。要表征這些具有完整免疫系統及相互作用細胞組分的模型,需要采用多重標記分析技術。我們展示了一種基于人工智能的空間蛋白質組學方法,用于研究小鼠癌組織中的腫瘤-免疫互作機制。
未經治療腫瘤的空間圖譜分析可呈現腫瘤免疫結構的整體特征,有助于理解治療反應。具有免疫活性的小鼠模型對于識別腫瘤發生發展過程中免疫依賴性事件至關重要。要表征這些具有完整免疫系統及相互作用細胞組分的模型,需要采用多重標記分析技術。我們展示了一種基于人工智能的空間蛋白質組學方法,用于研究小鼠癌組織中的腫瘤-免疫互作機制。
核心發現:
- 了解 Cell DIVE 多重成像技術與 Cell Signaling Technology 經 IHC 驗證抗體的結合如何實現多種細胞類型的精準檢測。
- 探討腫瘤細胞代謝與免疫抑制機制在癌癥進展中的作用。
- 探索如何利用 Aivia 進行 AI 引導的圖像分析以簡化空間蛋白質組學研究。
- 識別特定免疫細胞亞型、其相對于腫瘤區域的分布及相互作用,從而更深入地理解驅動腫瘤進展和治療反應的復雜動態機制。
01 多重成像與人工智能分析的整合應用
本研究探討了癌癥進展與治療過程中免疫系統與腫瘤細胞之間復雜的相互作用關系。Cell DIVE 多重成像技術能在單張組織切片上檢測數十種生物標志物,提供展示各類細胞類型的全組織分子圖譜。Cell Signaling Technology 提供的 IHC 驗證抗體與 Cell DIVE 完美兼容,可增強組織樣本中各類細胞的檢測與表型分析。Aivia 軟件的整合通過 AI 引導的分割、表型分析和聚類分析簡化了空間生物學工作流程。以 4T1 小鼠腫瘤模型為例的研究證實了該空間蛋白質組學工作流程的實用性,揭示了腫瘤內部異質性免疫景觀及復雜的空間關系。
通過 Aivia 平臺進行 AI 輔助分割與表型分析,我們對 Cell DIVE 多重全組織圖像中所有標志物的表達特征進行了系統表征(圖 1)。
 圖 1:使用 Aivia 進行多重圖像分析。A. Cell DIVE 全組織多重圖像及局部放大面板。B. 通過 Aivia 的多重細胞檢測方案實現細胞膜與核膜分割,將各細胞識別為獨立對象。C. AI 輔助表型分析鑒定各類細胞,包括免疫細胞、腫瘤細胞、成纖維細胞、內皮細胞及 Glut1 陽性細胞(可能屬于免疫或腫瘤細胞等不同類型)。
圖 1:使用 Aivia 進行多重圖像分析。A. Cell DIVE 全組織多重圖像及局部放大面板。B. 通過 Aivia 的多重細胞檢測方案實現細胞膜與核膜分割,將各細胞識別為獨立對象。C. AI 輔助表型分析鑒定各類細胞,包括免疫細胞、腫瘤細胞、成纖維細胞、內皮細胞及 Glut1 陽性細胞(可能屬于免疫或腫瘤細胞等不同類型)。02 腫瘤微環境對免疫細胞分布的影響
研究揭示了特定免疫細胞類型(如 T 細胞、巨噬細胞和樹突狀細胞)相對于腫瘤內缺氧與壞死區域的空間分布特征,表明腫瘤微環境對免疫細胞行為的影響及其對治療結局的潛在意義。
在同基因 4T1 小鼠腫瘤模型中,利用 Cell DIVE 和 Aivia 進行 AI 驅動的空間蛋白質組學分析,揭示了一個復雜多樣的免疫圖譜。總體而言,這項研究強調了多重成像技術與 AI 驅動分析相結合的重要性,以理解腫瘤微環境內的空間關系及相互作用,為開發更有效的癌癥療法提供了見解。
相關產品

CELL DIVE 超多標組織成像分析系統
 AI圖像分析軟件 Aivia
AI圖像分析軟件 AiviaCopyright(C) 1998-2025 生物器材網 電話:021-64166852;13621656896 E-mail:info@bio-equip.com