Nature文章分享:在體CRISPR screening技術助力發現增強CAR-T靶點
文章來源公眾號:生物快評 作者:bio2you
2025年9月24日, Nature發文《In vivo CRISPR screens identify modifiers of CAR T cell function in myeloma》,這篇文章是哈佛醫學院的Marcela V. Maus教授,她也是麻省總醫院細胞免疫治療項目的主任。

這里指的是CRISPR KO screening技術,簡單介紹一下:就是對一群T細胞轉導gRNA文庫,然后這群細胞中,某一些細胞亞群含有某一種基因敲除的表型。那么在給定一個篩選條件,讓能夠適應這種條件的、基因缺失的細胞群體,優勝劣汰出來。富集的T細胞在通過高通量測序分析是什么gRNA富集了,反推一下就知道是什么基因缺失了。
快速的結論
體外實驗中,敲除RASA2 和 SOCS1 可增強 T 細胞的擴增;而在體內實驗中,缺失PTPN2、ZC3H12A 和 RC3H1則賦予 CAR T 細胞早期的選擇性生長優勢。
值得注意的是,作者發現細胞周期調控因子——細胞周期蛋白依賴性激酶抑制因子 1B(CDKN1B) 是限制 CAR T 細胞在體內后期活性的重要因素。
敲除 CDKN1B可增強 BCMA CAR T 細胞對抗原的增殖和效應功能,從而顯著提高腫瘤清除率和整體生存率。
目前在Google Scholar檢索,去掉引用,共有57萬“crispr screening”信息。
文章的細節1)是什么CRISPR篩選系統?
所用的是Mario’ library(有不同類型的gRNA文庫),該文庫靶向的是135個基因敲除,這些基因在一定程度上可能有T細胞的功能相關,每個基因有8個gRNA。對照無意gRNA有100個(靶向基因間區)。
這個篩選體系靶向的基因有限,所以敲除的靶點肯定在這個范圍內,這限制了更大范圍的篩選。
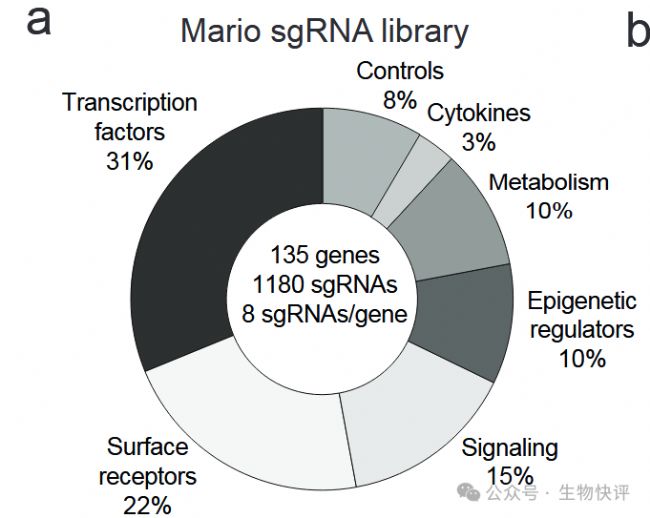
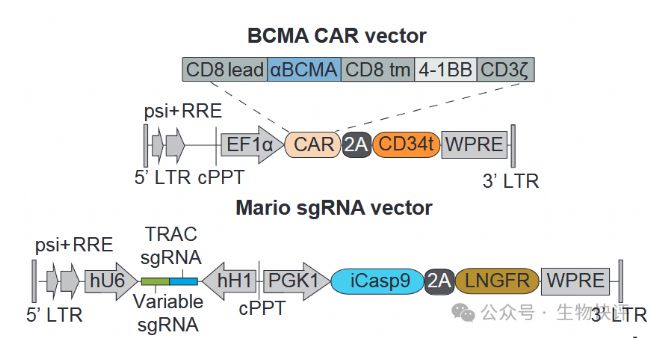
這個篩選所用的載體
載體1:靶向BCMA的CAR(4-1BBZ結構),這個CAR后面利用2A連接CD34t(短截體的CD34),該載體是表達CAR所用,CD34t是便于檢測CAR表達。
載體2:gRNA載體,該載體通過U6驅動gRNA轉錄,注意這里含有TRAC gRNA和靶向基因gRNA,TRAC gRNA是有兩個目的:其一是為了避免后續體內篩選的BCMA-CAR-T對宿主造成GvHD,其二是為了檢測和分選CD3陽性的T細胞,因為CD3陽性的T細胞即是未發生基因編輯的細胞。串聯PGK1啟動子驅動iCasp9表達,且iCasp9通過2A連接了一個篩選標記LNGFR,icasp9是一個誘導性的安全開關,這些和篩選無關,可以不要。
2)這個篩選怎么操作的?
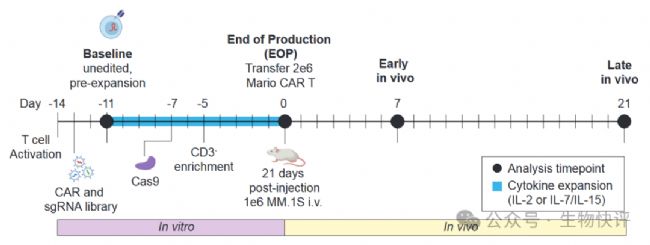
首先活化T細胞,然后制備含有BCMA-CAR的慢病毒和含有gRNA的文庫的慢病毒。
將上述慢病毒混合感染活化的T細胞,然后通過磁珠篩選去掉CD3陽性的T細胞,即得到了CD3陰性的經過文庫編輯后的T細胞。
然后這些細胞通過尾靜脈注射到含有MM.1S小鼠體內,在第21天在小鼠的骨髓中分離BCMA-CAR-T,最后通過測序關鍵敲除哪個基因的gRNA被富集了。
3)篩選得到什么基因敲除對于CAR-T比較好
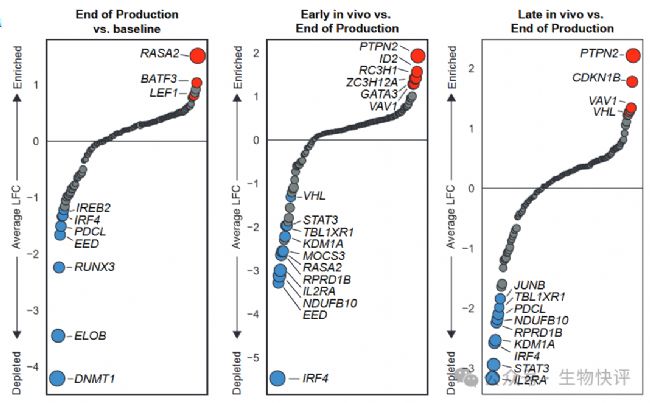
紅色是富集的gRNA的基因,即敲除帶來優勢的基因。
藍色為丟失的gRNA的基因,暗示敲除帶來不利影響。
因為這個是一個富集篩選,所以我們通常更加相信富集的敲除的基因更可靠。
題外話:基因敲除篩選真的就是一個“一網打盡”的系統嗎?
目前很多課題組做全基因組基因敲除篩選、過表達篩選,然后按照自己的觀察指標,進行基因富集。對得到靶點后再進行驗證,通常在加上單細胞測序的數據。
似乎“Screening”+“single-cell Seq”是發高分文章的套路。
實際上,某個領域的一些關鍵基因很多都在早期都被發現了。目前想通過大規模的篩選,很難得到你想要的“新”的靶點,大概率是對舊的機制、通路的縫縫補補。
測序和篩選的錢,大概率只是得到了一個文章的“頭”和“引子”。
其次你篩選得到的清單,非常困難得到一個不影響“其他功能”,只有好處的靶點。
最后很多人的篩選標準是“細胞增殖、細胞存活”,很多時候篩選得到了一些抗凋亡、促增長、或者是促癌基因,你所觀察到的指標只是“細胞長快了為富集”,而不是因為你的篩選壓力而富集。
怎么從別人的篩選中獲得自己關心的靶點呢
首先,一般文章會提供篩選的清單,頭部的靶點可能已經被作者重復,那么次要的靶點可以下載下來再看看。如果沒有現成的清單excel,直接下載測序數據,自己用軟件跑一下。
最后我們可以通過專利檢索一下,如果一年左右,作者沒有對發現靶點進行保護,大概率這些東西沒有什么實際作用。